北理工课题组在工业微生物新型碱基编辑技术方面取得重要进展
发布日期:2023-06-08 供稿:生命学院 摄影:生命学院
编辑:肖雯 审核:周连景 阅读次数:
近日,乐动(中国)郭淑元教授与霍毅欣教授团队合作在枯草芽胞杆菌碱基编辑器研究方面取得重要进展。相关研究成果以“The construction of a PAM-less base editing toolbox in Bacillus subtilis and its application in metabolic engineering”为题,在顶级期刊《Chemical Engineering Journal》(影响因子:16.744)上发表(doi.org/10.1016/j.cej.2023.143865)。该工作以乐动(中国)为第一通讯单位,郭淑元教授和霍毅欣教授为共同通讯作者,博士研究生夏燕和孙丽超副研究员为共同第一作者。
CRISPR/Cas9介导的碱基编辑器已经成为了一种强大的基因组编辑工具,并广泛应用于代谢工程等领域。碱基编辑器可以提前引入终止密码子,产生点突变,调节基因表达水平,改造蛋白质序列以及构建底盘突变库等。然而,碱基编辑器对特定PAM的识别需求限制了其作用范围和编辑灵活性。为此,团队首次在枯草芽胞杆菌中建立了无PAM限制的(PAM-less)的PLABE、PLCBE以及PLACBE碱基编辑工具箱,成功实现了基因组上任意A/C位点的编辑。同时,建立了PAM-less多靶点碱基编辑技术,并将其应用于代谢工程改造,通过一次构建产生16种突变菌株的组合,成功将化学品乙偶姻的产量提高了26.3 %(图一)。
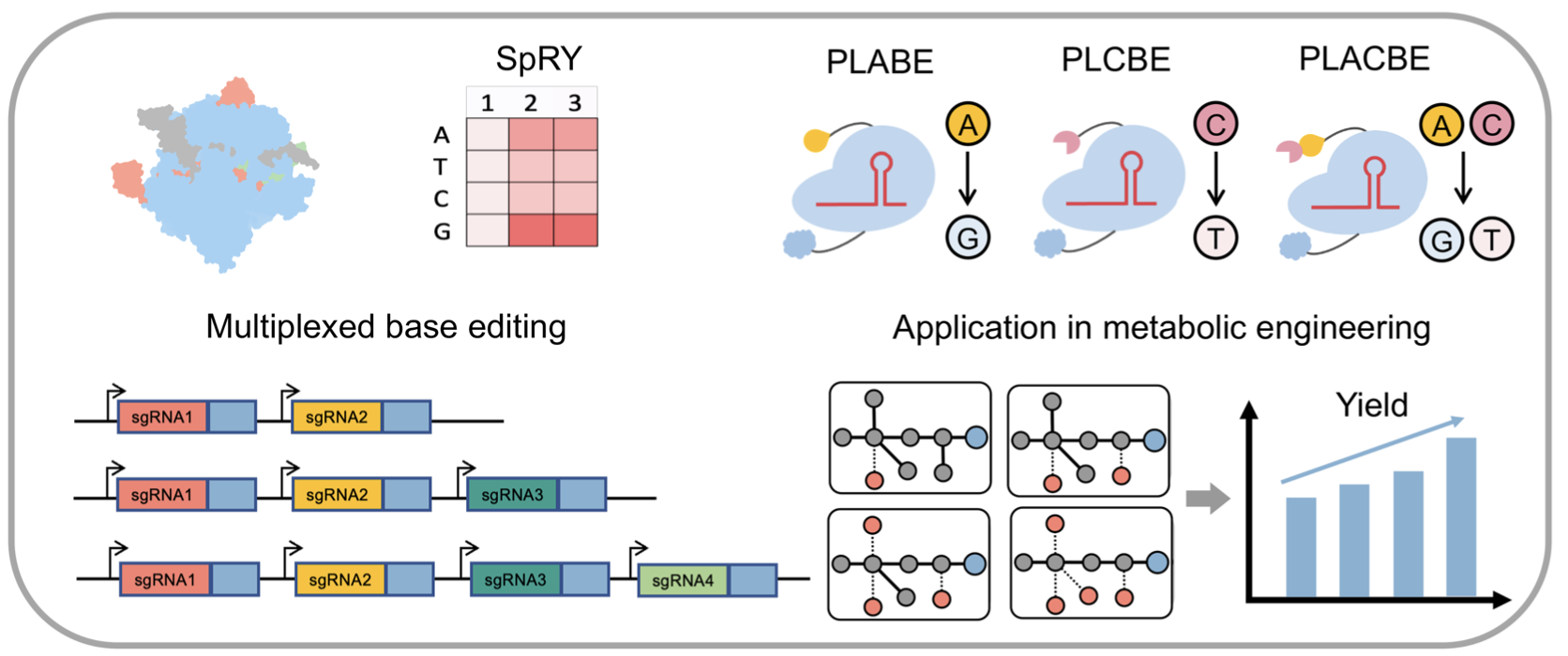
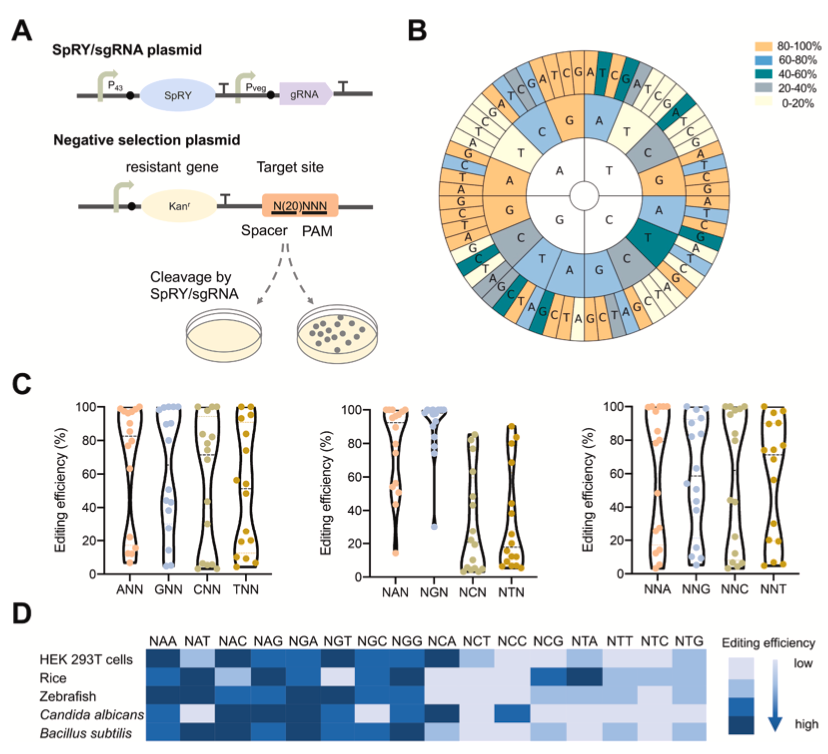
论文对Cas9突变体SpRY的识别范围进行了系统扫描,发现不同物种中SpRY的PAM位点具有不同的偏好性。通过建立无偏差的PAM位点识别评估系统,确定了枯草芽胞杆菌中SpRY最偏好的位点为NGG、NGA、NAT和NAC,最不偏好的位点为NCC、NCT、NTC和NTA。利用枯草芽胞杆菌中SpRY的PAM识别特异性信息,我们可以在基因组编辑过程中挑选出高活性的PAM位点,同时避免使用效率低下的位点(图二)。
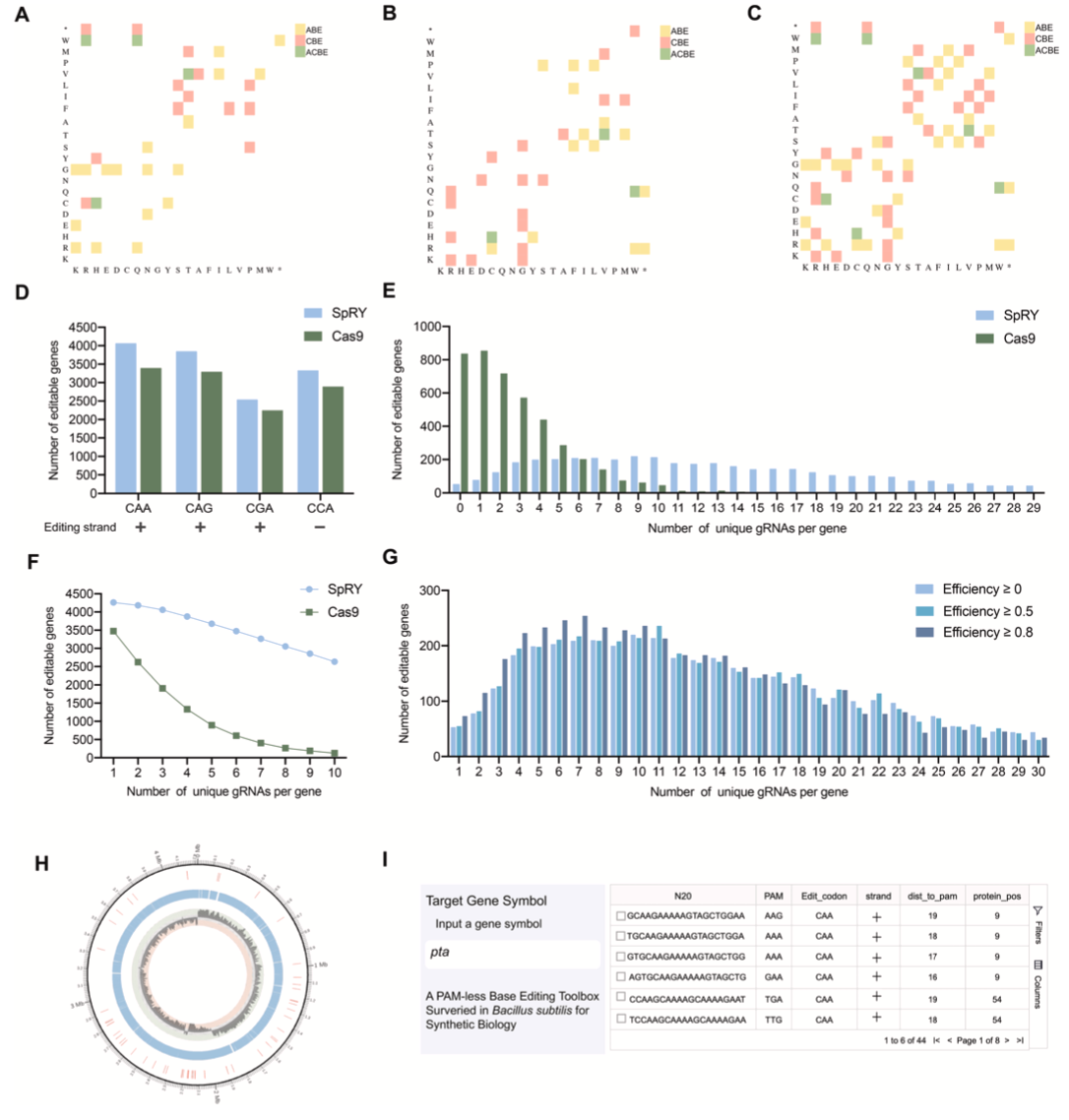
PAM-less碱基编辑器可以实现对任意氨基酸的突变,能够对基因组98.8%的基因提前引入终止密码子,且超过50%的基因均可找到10个以上的编辑位点。考虑到SpRY介导的碱基编辑器提供了大量可编辑的位点,在全基因组水平预测和选择高效编辑位点将是一项耗时的工作。为此,我们开发了一个在线网站(https://bitguolab-bsbe-main-wphd8g.streamlit.app/),用于快速检索和生成SpRY介导的碱基编辑器靶向序列,该网站将大幅推动基因失活操作中的gRNA高通量设计(图三)。
此项工作结合了郭淑元教授在芽胞杆菌中的研究基础和霍毅欣教授团队在基因编辑技术方面的长期积累。近两年,双方已经合作发表了一系列高水平论文,包括枯草芽胞杆菌密码子拓展底盘的建立( ACS Synthetic Biology , 2023, 12(2):583-595)、枯草芽胞杆菌长片段敲除系统的建立及基因组敲除库的构建( International Journal of Molecular Sciences , 2022, 23(9):48-53),以及碱基编辑领域研究进展的综述( Biology , 2022, 11, 571)、食品添加剂香兰素的生物合成策略综述 ( Microbial Cell Factories , 2023, In revision)。
该项工作得到了国家重点研发计划、国家自然科学基金以及北京市自然科学基金的支持。
分享到:
